Muy a menudo los mapas de plásmidos, especialmente los históricos que son dibujados a mano por un estudiante de doctorado olvidado hace mucho tiempo, son un rompecabezas. ¿Qué son estas flechas y cajas? ¿Por dónde empiezo? No te preocupes, tenemos un curso intensivo de introducción al descifrado de mapas de plásmidos.
Familiarizarse con Su Plásmido de Interés
Comencemos con un plásmido clásico: pBR3221. A menudo se usa como columna vertebral para vectores derivados porque tiene todas las características necesarias para una clonación exitosa (Figura 1). Como puede ver en el centro del mapa, el tamaño del plásmido linealizado es de 4361 pares de bases. Antes de comenzar a trabajar con cualquier plásmido, es aconsejable linealizarlo cortándolo con una enzima de restricción única para verificar que el tamaño anunciado sea aproximadamente el mismo que se espera.
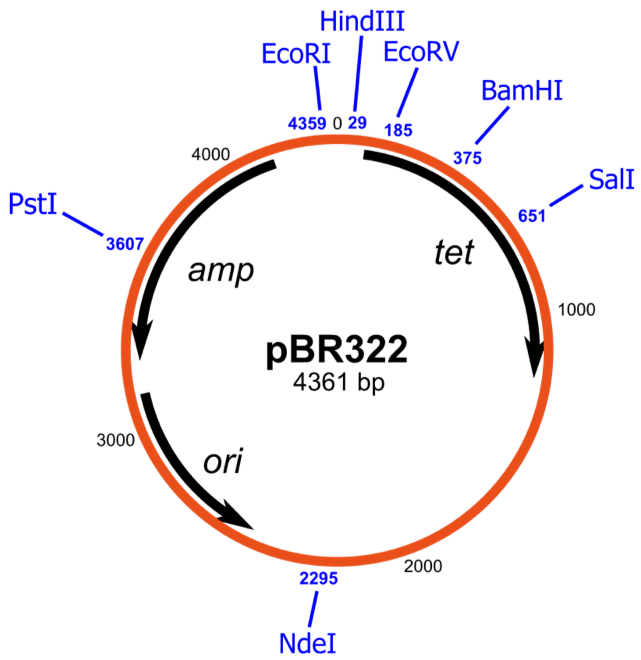
Figura 1. Esquema de pBR322. Imagen de Avacop & Yikrazuul a través de Wikimedia Commons
Las flechas negras muestran la dirección de la transcripción, que es esencial para la clonación. Si clona su gen de interés en el medio de otro gen, asegúrese de que ambos estén transcritos en la misma dirección. De lo contrario, el promotor nativo puede interferir con la expresión génica.
¿Qué Significa «Ori»?
En las bases de datos de secuencias de genes, tales como Entrez-PubMed, las secuencias de plásmidos se diagraman como secuencias lineales a partir de los ori. «Ori» que significa el origen de la replicación del plásmido. Hagas lo que hagas, no lo cambies! Una vez que un plásmido es incapaz de replicarse, es inútil.
La otra cosa a recordar sobre los ori es que los plásmidos con el mismo origen a menudo son incompatibles. Esto significa que no podrá mantener dos vectores derivados de pBR323 en una célula, incluso si tienen genes para diferentes resistencias a antibióticos. El ori pBR322 también se usa en pUC18, que es la segunda columna vertebral más común utilizada en vectores eucarióticos.
¿Dónde puedo Encontrar los Sitios de Restricción?
Los sitios de restricción para las enzimas correspondientes se muestran como líneas verticales con la posición de los nucleótidos iniciales. Los sitios deben ser únicos, pero vale la pena comprobarlos, ya que los vectores derivados a menudo contienen secuencias olvidadas adicionales.
¿Qué Pasa Con Los Genes De Resistencia A Los Antibióticos?
pBR322 tiene dos genes de resistencia a los antibióticos: tet (resistencia a la tetraciclina) y amp (resistencia a la ampicilina). Estos genes codifican una bomba de eflujo (tetR) y beta-lactamasa (ampR) para excretar tetraciclina y ampicilina de la célula, respectivamente. Tet y amp se leen en diferentes direcciones.
Tenga en cuenta que la enzima beta-lactamasa no es específica para desintoxicar los antibióticos derivados de la penicilina. Por lo tanto, incluso si tiene dos plásmidos con diferentes orígenes de replicación, no podrá seleccionar dos plásmidos al mismo tiempo si uno expresa un gen de resistencia a la meticilina y el otro expresa un gen de resistencia a la ampicilina.
Cuando use sitios de enzimas de restricción para clonar su gen de interés en su plásmido, tenga cuidado de observar qué sitios están dentro de su gen de resistencia a los antibióticos. Por ejemplo, los cortes de PvuI en el medio de AmpR y los cortes de BamHI en el medio de TetR. Y, como todos sabemos, la interrupción en un gen conducirá a la inactivación de la función génica, en este caso, la resistencia a los antibióticos.
¿Cómo se Inicia y Detiene la Replicación?
Además de los genes, los plásmidos a menudo incluyen promotores de transcripción y terminadores derivados de fagos de E. coli. Los promotores de los fagos SP6 y T7 se utilizan a menudo para la amplificación de ARN in vitro. Requieren polimerasas fágicas y, por lo tanto, son inactivas in vivo.
A continuación se muestra un mapa de pTLNX , un vector de expresión de ovocitos Xenopus (Figura 2). Además de los conocidos genes de origen pBR322 y resistencia a los antibióticos AmpR y CmR (resistencia al cloranfenicol), también hay promotores de SP6 y lacUV presentes. Aguas abajo del promotor SP6, el terminador rrnBT2 permite la terminación eficiente de genes clonados en el sitio de clonación múltiple 2 (Figura 2).
El vector pTLNX también tiene un gen para la selección de plásmidos (ccdB), junto con la señal de localización nuclear del virus SV40 y Xenopus globine 3′ UTR, que permite altos niveles de expresión de genes clonados.
Conozca sus fuentes
Los mapas caseros a menudo no son confiables, ya que omiten características «sin importancia» que pueden ser críticas para su experimento. Si necesita un mapa vectorial, es mejor usar repositorios de mapas conocidos como Addgene, Entrez-PubMed y sitios de empresas que venden su vector de interés.

Figura 2. Esquema de pTLNX. Imagen de Addgene.
Los mapas de plásmidos siempre están evolucionando, por lo que es probable que sus contribuciones se dejen para sus futuros colegas. Por favor, incluya tantos detalles como sea posible en su mapa! Feliz lectura y dibujo de mapas.
Fuentes:
- Balbás P, Soberón X, Merino E, Zurita M, Lomeli H, Valle F, Flores N, Bolívar F (1986). «Plasmid vector pBR322 and its special-purpose derivatives-a review»(en inglés). Gen. 50 (1-3): 3–40. doi: 1016/0378-1119 (86)90307-0.
- Geertsma ER, Dutzler R. (2011). Una herramienta de clonación versátil y eficiente de alto rendimiento para biología estructural. 50(15):3272-8. doi: 10.1021 / bi200178z
¿Esto te ha ayudado? Entonces, por favor, comparta con su red.