Très souvent, les cartes des plasmides, en particulier les cartes historiques dessinées à la main par un doctorant oublié depuis longtemps, sont un casse-tête. Quelles sont ces flèches et ces boîtes? Par où commencer ? Ne vous inquiétez pas, nous avons un cours intensif d’introduction au déchiffrement des cartes plasmidiques.
Se familiariser avec Son Plasmide d’intérêt
Commençons par un plasmide classique : pBR3221. Il est souvent utilisé comme épine dorsale pour les vecteurs dérivés car il possède toutes les fonctionnalités nécessaires à un clonage réussi (Figure 1). Comme vous le voyez sur le centre de la carte, la taille du plasmide linéarisé est de 4361 paires de bases. Avant de commencer à travailler avec un plasmide, il est conseillé de le linéariser en le coupant avec une enzyme de restriction unique pour vérifier que la taille annoncée est à peu près la même que prévue.
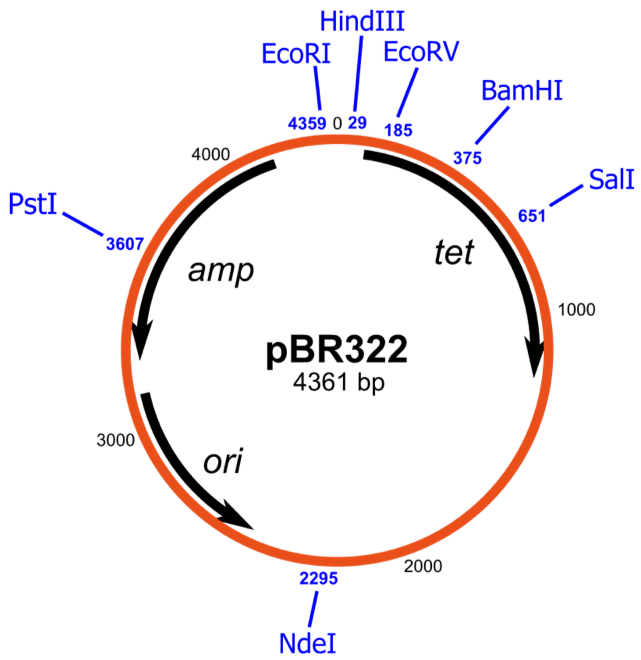
Figure 1. Schéma de pBR322. Image par Avacop & Yikrazuul via Wikimedia Commons
Les flèches noires indiquent la direction de la transcription, essentielle au clonage. Si vous clonez votre gène d’intérêt au milieu d’un autre gène, assurez-vous que les deux sont transcrits dans la même direction. Sinon, le promoteur natif peut interférer avec l’expression de votre gène.
Que signifie « Ori »?
Dans les bases de données de séquences de gènes, de telles séquences plasmidiques Entrez-PubMed sont schématisées en séquences linéaires à partir de l’ori. « Ori » signifie l’origine de la réplication plasmidique. Quoi que vous fassiez, ne le changez pas! Une fois qu’un plasmide est incapable de se répliquer, il est inutile.
L’autre chose à retenir à propos d’ori est que les plasmides de même origine sont souvent incompatibles. Cela signifie que vous ne pourrez pas maintenir deux vecteurs dérivés du pBR323 dans une cellule même s’ils ont des gènes pour différentes résistances aux antibiotiques. Le pBR322 ori est également utilisé dans pUC18, qui est le deuxième squelette le plus couramment utilisé dans les vecteurs eucaryotes.
Où Puis-je Trouver les Sites de restriction ?
Les sites de restriction des enzymes correspondantes sont représentés sous forme de lignes verticales avec la position des nucléotides de départ. Les sites doivent être uniques, mais il est utile de les vérifier, car les vecteurs dérivés contiennent souvent des séquences oubliées supplémentaires.
Qu’En Est-Il Des Gènes De Résistance Aux Antibiotiques?
pBR322 possède deux gènes de résistance aux antibiotiques: tet (résistance à la tétracycline) et amp (résistance à l’ampicilline). Ces gènes codent une pompe d’efflux (tetR) et une bêta-lactamase (ampR) pour excréter respectivement la tétracycline et l’ampicilline de la cellule. Tet et amp sont lus dans des directions différentes.
Gardez à l’esprit que l’enzyme bêta-lactamase n’est pas spécifique des antibiotiques détoxifiants dérivés de la pénicilline. Ainsi, même si vous avez deux plasmides d’origines de réplication différentes, vous ne pourrez pas sélectionner deux plasmides en même temps si l’un exprime un gène de résistance à la méthicilline et l’autre un gène de résistance à l’ampicilline.
Lorsque vous utilisez des sites d’enzymes de restriction pour cloner votre gène d’intérêt dans votre plasmide, veillez à déterminer quels sites relèvent de votre gène de résistance aux antibiotiques. Par exemple, PvuI coupe au milieu de l’AmpR et BamHI coupe au milieu du TetR. Et, comme nous le savons tous, la perturbation d’un gène entraînera l’inactivation de la fonction génique – dans ce cas, la résistance aux antibiotiques.
Comment la réplication Démarre-t-Elle et s’arrête-t-elle ?
En plus des gènes, les plasmides comprennent souvent des promoteurs et des terminateurs de transcription dérivés de phages d’E.coli. Les promoteurs des phages SP6 et T7 sont souvent utilisés pour l’amplification de l’ARN in vitro. Ils nécessitent des polymérases phagiques et sont donc inactifs in vivo.
Voici une carte de pTLNX, un vecteur d’expression des ovocytes de Xénope (Figure 2). En plus des gènes familiers d’origine pBR322 et de résistance aux antibiotiques AmpR et CmR (résistance au chloramphénicol), des promoteurs SP6 et lacUV sont également présents. En aval du promoteur SP6, le terminateur rrnBT2 permet une terminaison efficace des gènes clonés dans le site de clonage multiple 2 (Figure 2).
Le vecteur pTLNX possède également un gène de sélection des plasmides (ccdB), ainsi que le signal de localisation nucléaire du virus SV40 et la globine de xénope 3′ UTR, qui permettent des niveaux d’expression élevés de gènes clonés.
Connaissez vos sources
Les cartes faites maison ne sont souvent pas fiables car elles omettent des fonctionnalités « sans importance » qui peuvent être critiques pour votre expérience. Si vous avez besoin d’une carte vectorielle, il est préférable d’utiliser des référentiels cartographiques connus tels que Addgene, Entrez-PubMed et les sites des entreprises qui vendent votre vecteur d’intérêt.

Figure 2. Schéma de pTLNX. Image de Addgene.
Les cartes plasmidiques sont en constante évolution, il est donc probable que vos contributions seront laissées à vos futurs collègues. Veuillez inclure autant de détails que possible sur votre carte! Bonne lecture et dessin de carte.
Sources:
- Balbás P, Soberón X, Merino E, Zurita M, Lomeli H, Valle F, Flores N, Bolivar F (1986). « Plasmide vector pBR322 et ses dérivés à usage spécial – une revue ». Gène. 50 (1-3): 3–40. doi: 1016/0378- 1119 (86) 90307-0.
- Geertsma ER, Dutzler R. (2011). Un outil de clonage à haut débit polyvalent et efficace pour la biologie structurale. 50(15):3272-8. doi: 10.1021 / bi200178z
Cela vous a-t-il aidé ? Ensuite, veuillez partager avec votre réseau.