Molto spesso le mappe plasmidiche, specialmente quelle storiche che sono disegnate a mano da uno studente di dottorato dimenticato da tempo, sono un puzzle. Quali sono queste frecce e scatole? Da dove comincio? Non preoccuparti, abbiamo un’introduzione al corso accelerato per decifrare le mappe plasmidiche.
Familiarizzare con il tuo plasmide di interesse
Iniziamo con un plasmide classico: pBR3221. È spesso usato come spina dorsale per i vettori derivati perché ha tutte le caratteristiche necessarie per una clonazione riuscita (Figura 1). Come vedi dal centro della mappa, la dimensione del plasmide linearizzato è 4361 coppie di basi. Prima di iniziare a lavorare con qualsiasi plasmide, è consigliabile linearizzarlo tagliando con un enzima di restrizione unico per verificare che la dimensione pubblicizzata sia all’incirca la stessa del previsto.
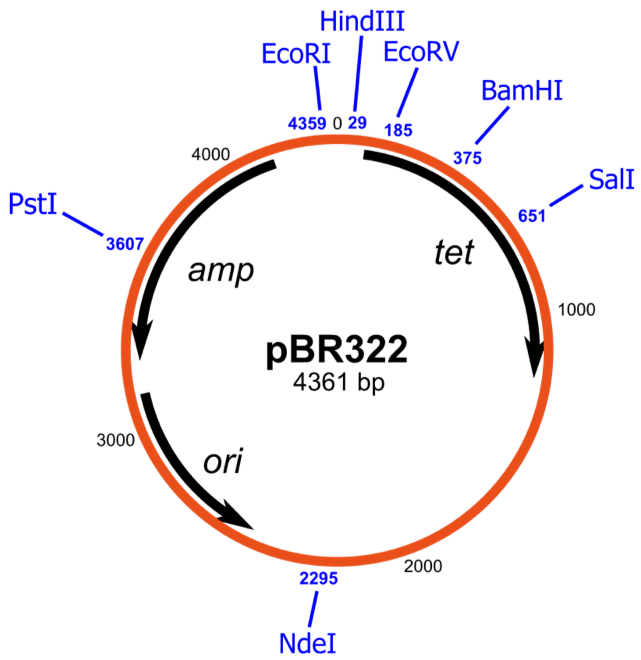
Figura 1. Schema di pBR322. Immagine di Avacop & Yikrazuul via Wikimedia Commons
Le frecce nere mostrano la direzione della trascrizione, che è essenziale per la clonazione. Se cloni il tuo gene di interesse in un mezzo di un altro gene, assicurati che entrambi siano trascritti nella stessa direzione. Altrimenti, il promotore nativo può interferire con la tua espressione genica.
Cosa significa “Ori”?
Nei database di sequenze geniche, tali sequenze di plasmidi Entrez-PubMed sono diagrammate come sequenze lineari a partire dagli ori. “Ori” indica l’origine della replicazione plasmidica. Qualunque cosa tu faccia, non cambiarla! Una volta che un plasmide non è in grado di replicarsi, è inutile.
L’altra cosa da ricordare su ori è che i plasmidi con la stessa origine sono spesso incompatibili. Ciò significa che non sarà in grado di mantenere due vettori derivati da pBR323 in una cellula anche se hanno geni per diverse resistenze antibiotiche su di essi. Il pbr322 ori inoltre è usato in pUC18, che è la seconda spina dorsale più comune utilizzata nei vettori eucariotici.
Dove posso trovare i siti di restrizione?
I siti di restrizione per gli enzimi corrispondenti sono mostrati come linee verticali con la posizione dei nucleotidi di partenza. I siti dovrebbero essere unici, ma vale la pena controllare, poiché i vettori derivati spesso contengono sequenze aggiuntive dimenticate.
Che dire dei geni di resistenza agli antibiotici?
pBR322 ha due geni di resistenza agli antibiotici: tet (resistenza alle tetracicline) e amp (resistenza all’ampicillina). Questi geni codificano una pompa di efflusso (tetR) e beta-lattamasi (ampR) per espellere tetraciclina e ampicillina dalla cellula, rispettivamente. Tet e amp vengono letti in direzioni diverse.
Tenere presente che l’enzima beta-lattamasi non è specifico nella disintossicazione degli antibiotici derivati dalla penicillina. Quindi, anche se hai due plasmidi con origini diverse di replicazione, non sarai in grado di selezionare due plasmidi allo stesso tempo se uno esprime un gene di resistenza alla meticillina e l’altro esprime un gene di resistenza all’ampicillina.
Quando si utilizzano siti enzimatici di restrizione per clonare il gene di interesse nel plasmide, fare attenzione a guardare quali siti rientrano nel gene di resistenza agli antibiotici. Ad esempio, PvuI taglia nel mezzo di AmpR e BAMHI taglia nel mezzo di TetR. E, come tutti sappiamo, l’interruzione di un gene porterà all’inattivazione della funzione genica – in questo caso, resistenza agli antibiotici.
Come si avvia e si arresta la replica?
Oltre ai geni, i plasmidi spesso includono promotori di trascrizione e terminatori derivati da fagi di E. coli. I promotori dei fagi SP6 e T7 sono spesso utilizzati per l’amplificazione in vitro dell’RNA. Richiedono polimerasi di fagi e sono quindi inattivi in vivo.
Di seguito è riportata una mappa di pTLNX, un vettore di espressione degli ovociti Xenopus (Figura 2). Oltre alla familiare origine pBR322 e ai geni di resistenza agli antibiotici AmpR e CmR (resistenza al cloramfenicolo), sono presenti anche promotori SP6 e lacUV. A valle del promotore SP6, il terminatore rrnBT2 consente la terminazione efficiente dei geni clonati nel sito di clonazione multipla2 (Figura 2).
Il vettore pTLNX ha anche un gene per la selezione plasmidica (ccdB), insieme al virus SV40 nuclear localization signal e Xenopus globine 3′ UTR, che consente alti livelli di espressione di geni clonati.
Conosci le tue fonti
Le mappe fatte in casa sono spesso inaffidabili in quanto omettono funzionalità “non importanti” che potrebbero essere critiche per il tuo esperimento. Se hai bisogno di una mappa vettoriale, è meglio usare repository di mappe noti come Addgene, Entrez-PubMed e siti di aziende che vendono il tuo vettore di interesse.

Figura 2. Schema di pTLNX. Immagine da Addgene.
Le mappe plasmidiche sono in continua evoluzione, quindi è probabile che i tuoi contributi saranno lasciati ai tuoi futuri colleghi. Si prega di includere il maggior numero possibile di dettagli nella mappa! Buona lettura e disegno della mappa.
Fonti:
- Balbás P, Soberón X, Merino E, Zurita M, Lomeli H, Valle F, Flores N, Bolivar F (1986). “Plasmid vector pBR322 and its special-purpose derivatives-a review”. Gene. 50 (1-3): 3–40. doi:1016/0378-1119 (86)90307-0.
- Geertsma ER, Dutzler R. (2011). Uno strumento di clonazione versatile ed efficiente ad alto rendimento per la biologia strutturale. 50(15):3272-8. doi: 10.1021 / bi200178z
Questo ti ha aiutato? Quindi si prega di condividere con la rete.