bardzo często mapy plazmidowe, zwłaszcza historyczne, które są ręcznie rysowane przez dawno zapomnianego Doktoranta, są zagadką. Co to za Strzałki i pudła? Od czego mam zacząć? Nie martw się, mamy kurs wprowadzający do rozszyfrowania map plazmidowych.
zapoznanie się z interesującym Cię plazmidem
Zacznijmy od klasycznego plazmidu: pBR3221. Jest często używany jako szkielet dla wektorów pochodnych, ponieważ posiada wszystkie cechy potrzebne do udanego klonowania (Rysunek 1). Jak widać z centrum mapy, rozmiar zlinearyzowanego plazmidu wynosi 4361 par zasad. Przed rozpoczęciem pracy z dowolnym plazmidem wskazane jest linearyzowanie go poprzez cięcie za pomocą unikalnego enzymu restrykcyjnego, aby sprawdzić, czy reklamowany rozmiar jest w przybliżeniu taki sam, jak oczekiwano.
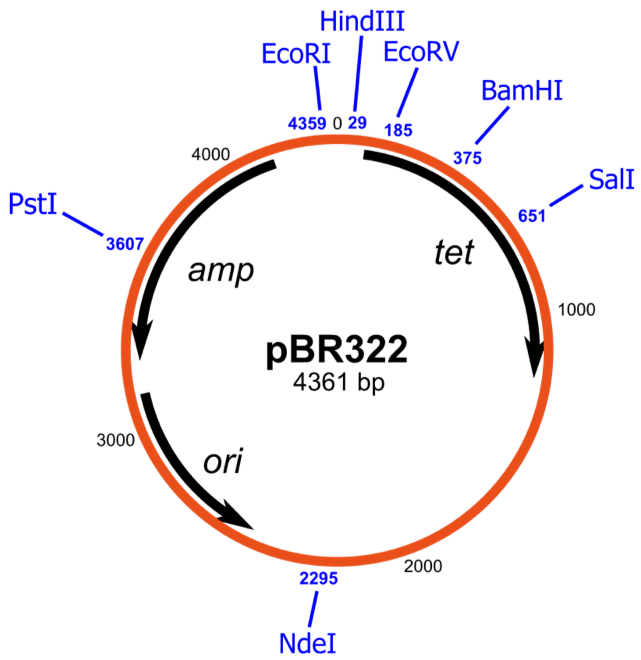
Rysunek 1. Schemat pbr322. Zdjęcie Avacop & yikrazuul via Wikimedia Commons
czarne strzałki pokazują kierunek transkrypcji, który jest niezbędny do klonowania. Jeśli sklonujesz interesujący Cię gen w środku innego genu, upewnij się, że oba są transkrybowane w tym samym kierunku. W przeciwnym razie natywny promotor może zakłócać ekspresję genów.
Co Oznacza „Ori”?
w bazach sekwencji genów, takich jak Entrez-PubMed, sekwencje plazmidowe są diagramowane jako sekwencje liniowe, począwszy od ori. „Ori” oznacza pochodzenie replikacji plazmidów. Cokolwiek zrobisz, nie zmieniaj tego! Gdy plazmid nie jest w stanie replikować, jest bezużyteczny.
inną rzeczą do zapamiętania o ori jest to, że plazmidy o tym samym pochodzeniu są często niekompatybilne. Oznacza to, że nie będziesz w stanie utrzymać dwóch wektorów pochodnych pBR323 w jednej komórce, nawet jeśli mają one geny dla różnych oporności na antybiotyki. Pbr322 ori jest również używany w pUC18, który jest drugim najczęściej stosowanym szkieletem stosowanym w wektorach eukariotycznych.
Gdzie mogę znaleźć strony z ograniczeniami?
miejsca restrykcyjne dla odpowiednich enzymów są pokazane jako pionowe linie z położeniem początkowych nukleotydów. Strony powinny być unikalne, ale opłaca się to sprawdzić, ponieważ wektory pochodne często zawierają dodatkowe zapomniane sekwencje.
A Co Z Genami Oporności Na Antybiotyki?
pBR322 ma dwa geny oporności na antybiotyki: tet (oporność na tetracykliny) i amp (oporność na ampicylinę). Geny te kodują pompę wypływu (tetR) i beta-laktamazę (ampR) do wydalania tetracykliny i ampicyliny z komórki, odpowiednio. Tet i amp są odczytywane w różnych kierunkach.
należy pamiętać, że enzym beta-laktamaza nie jest specyficzny w detoksykacji antybiotyków pochodzących z penicyliny. Więc nawet jeśli masz dwa plazmidy o różnym pochodzeniu replikacji, nie będziesz w stanie wybrać dwóch plazmidów w tym samym czasie, jeśli jeden będzie wykazywał Gen oporności na metycylinę, a drugi genu oporności na ampicylinę.
podczas używania miejsc enzymu restrykcyjnego do klonowania interesującego genu do plazmidu, należy uważać, aby sprawdzić, które miejsca należą do genu oporności na antybiotyki. Na przykład PvuI tnie w środku AmpR, a BamHI tnie w środku TetR. I, jak wszyscy wiemy, zakłócenia w genie doprowadzi do inaktywacji funkcji genu – w tym przypadku, oporność na antybiotyki.
jak rozpoczyna się i zatrzymuje replikacja?
oprócz genów plazmidy często zawierają promotory transkrypcji i terminatory pochodzące z fagów E. coli. Promotory z fagów SP6 i T7 są często używane do amplifikacji RNA in vitro. Wymagają one polimeraz fagowych i dlatego są nieaktywne in vivo.
poniżej znajduje się Mapa pTLNX, wektora ekspresyjnego Xenopus oocytu (Rysunek 2). Oprócz znanych genów pochodzenia pBR322 i oporności na antybiotyki AmpR i CmR (oporność na chloramfenikol), istnieją również promotory SP6 i lacUV. Poniżej promotora SP6, terminator rrnbt2 umożliwia skuteczne zakończenie genów sklonowanych w miejscu wielokrotnego klonowania2 (fig.2).
wektor pTLNX ma również gen do selekcji plazmidów (ccdB), wraz z sygnałem lokalizacji jądrowej wirusa SV40 i UTR Xenopus globine 3′, który pozwala na wysoki poziom ekspresji klonowanych genów.
Poznaj swoje źródła
domowe mapy są często zawodne, ponieważ pomijają „nieistotne” funkcje, które mogą być krytyczne dla Twojego eksperymentu. Jeśli potrzebujesz mapy wektorowej, lepiej użyć znanych repozytoriów map, takich jak Addgene, Entrez-PubMed i witryn firm, które sprzedają interesujący Cię wektor.

Rysunek 2. Schemat pTLNX. Zdjęcie z Addgene.
Mapy plazmidów stale się rozwijają, więc prawdopodobnie Twój wkład zostanie pozostawiony przyszłym kolegom. Proszę podać jak najwięcej szczegółów na mapie! Miłego czytania i rysowania map.
:
- Balbás P, Soberón X, Merino E, Zurita M, Lomeli H, Valle F, Flores N, Bolivar F (1986). Plazmid vector pBR322 and its special-purpose derivatives–a review (ang.). Gene. 50 (1-3): 3–40. doi: 1016/0378-1119 (86)90307-0.
- Geertsma ER, Dutzler R. (2011). Wszechstronne i wydajne narzędzie do klonowania o wysokiej wydajności do biologii strukturalnej. 50(15):3272-8. doi: 10.1021 / bi200178z
pomogło ci to? Następnie podziel się z siecią.