muitas vezes mapas de plasmídeos, especialmente os históricos desenhados à mão por um estudante de Doutorado há muito esquecido, são um quebra-cabeça. Quais são essas setas e caixas? Por onde começo? Não se preocupe, temos uma introdução de curso intensivo para decifrar mapas de plasmídeos.
familiarizando – se com o seu plasmídeo de interesse
vamos começar com um plasmídeo clássico: pBR3221. É frequentemente usado como um backbone para vetores derivados porque possui todos os recursos necessários para uma clonagem bem-sucedida (Figura 1). Como você vê no centro do mapa, o tamanho do plasmídeo linearizado é de 4361 pares de bases. Antes de começar a trabalhar com qualquer plasmídeo, é aconselhável linearizá-lo cortando com uma enzima de restrição única para verificar se o tamanho anunciado é aproximadamente o mesmo esperado.
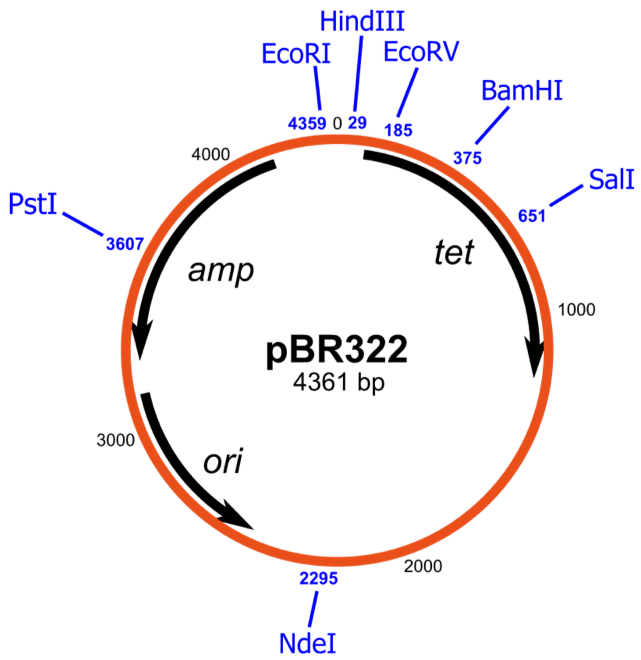
Figura 1. Esquema do pBR322. Imagem por Avacop & Yikrazuul via Wikimedia Commons
as setas pretas mostram a direção da transcrição, que é essencial para a clonagem. Se você clonar seu gene de interesse em um meio de outro gene, certifique-se de que ambos sejam transcritos na mesma direção. Caso contrário, o promotor nativo pode interferir na expressão gênica.
O Que Significa “Ori”?
em bancos de dados de sequência gênica, tais sequências de plasmídeos Entrez-PubMed são diagramadas como sequências lineares a partir do ori. “Ori” significa a origem da replicação do plasmídeo. Faça o que fizer, não mude! Uma vez que um plasmídeo é incapaz de se replicar, é inútil.
a outra coisa a lembrar sobre ori é que os plasmídeos com a mesma origem são frequentemente incompatíveis. Isso significa que você não será capaz de manter dois vetores derivados de pBR323 em uma célula, mesmo que tenham genes para diferentes resistências a antibióticos. O pBR322 ori também é usado em puc18, que é o segundo backbone mais comum usado em vetores eucarióticos.
Onde posso encontrar os Sites de restrição?
os locais de restrição para enzimas correspondentes são mostrados como linhas verticais com a posição dos nucleotídeos iniciais. Os sites devem ser únicos, mas vale a pena verificar, pois os vetores derivados geralmente contêm sequências esquecidas adicionais.
E Quanto Aos Genes De Resistência Aos Antibióticos?
o pBR322 possui dois genes de Resistência a antibióticos: tet (resistência à tetraciclina) e amp (resistência à ampicilina). Esses genes codificam uma bomba de efluxo (tetR) e beta-lactamase (ampR) para excretar tetraciclina e ampicilina da célula, respectivamente. TET e amp são lidos em diferentes direções.
tenha em mente que a enzima beta-lactamase não é específica na desintoxicação de antibióticos derivados da penicilina. Portanto, mesmo se você tiver dois plasmídeos com origens diferentes de replicação, não poderá selecionar dois plasmídeos ao mesmo tempo se um expressar um gene de resistência à meticilina e o outro expressar um gene de resistência à ampicilina.
ao usar locais de enzimas de restrição para clonar seu gene de interesse em seu plasmídeo, tome cuidado para ver quais locais se enquadram em seu gene de Resistência a antibióticos. Por exemplo, PvuI corta no meio de AmpR, e BamHI corta no meio de TetR. E, como todos sabemos, a interrupção em um gene levará à inativação da função gênica – neste caso, resistência a antibióticos.
como a replicação inicia e para?
além dos genes, os plasmídeos geralmente incluem promotores de transcrição e terminadores derivados de fagos de E. coli. Promotores de fagos SP6 e T7 são freqüentemente usados para amplificação de RNA in vitro. Eles requerem polimerases de fagos e, portanto, são inativos in vivo.
abaixo está um mapa de pTLNX, um vetor de expressão de oócitos Xenopus (Figura 2). Além dos familiares genes de origem pBR322 e resistência a antibióticos AmpR e CmR (resistência ao cloranfenicol), também estão presentes promotores de SP6 e lacUV. A jusante do promotor SP6, o terminador rrnbt2 permite a terminação eficiente de genes clonados no local de clonagem múltipla 2 (Figura 2).
o vetor pTLNX também possui um gene para seleção de plasmídeos (ccdB), juntamente com o sinal de localização nuclear do vírus SV40 e Xenopus globine 3′ UTR, que permite altos níveis de expressão de genes clonados.
Conheça suas fontes
mapas caseiros geralmente não são confiáveis, pois omitem recursos “sem importância” que podem ser críticos para o seu experimento. Se você precisar de um mapa vetorial, é melhor usar repositórios de mapas conhecidos, como Addgene, Entrez-PubMed e sites de empresas que vendem seu Vetor de interesse.

Figura 2. Esquema do pTLNX. Imagem de Addgene.
os mapas de plasmídeos estão sempre evoluindo, por isso é provável que suas contribuições sejam deixadas para seus futuros colegas. Por favor, inclua o máximo de detalhes possível no seu mapa! Feliz mapa de leitura e desenho.
Fontes:
- Balbás P, Soberón X, Merino E, Zurita M, Lomeli H, Valle F, Flores N, Bolivar F (1986). “Plasmid vector pBR322 and its special-purpose derivatives–a review” (em inglês). Gene. 50 (1-3): 3–40. doi: 1016/0378-1119 (86)90307-0.
- Geertsma ER, Dutzler R. (2011). Uma ferramenta versátil e eficiente da clonagem da alto-taxa de transferência para a biologia estrutural. 50(15):3272-8. doi: 10.1021 / bi200178z
isto ajudou-te? Então, por favor, compartilhe com sua rede.