Sehr oft sind Plasmidkarten, insbesondere historische, die von einem längst vergessenen Doktoranden von Hand gezeichnet wurden, ein Rätsel. Was sind diese Pfeile und Boxen? Wo fange ich an? Keine Sorge, wir haben einen Crashkurs Einführung in die Entschlüsselung Plasmid Karten.
Sich mit Ihrem interessierenden Plasmid vertraut machen
Beginnen wir mit einem klassischen Plasmid: pBR3221. Es wird häufig als Rückgrat für abgeleitete Vektoren verwendet, da es über alle Funktionen verfügt, die für eine erfolgreiche Klonierung erforderlich sind (Abbildung 1). Wie Sie in der Kartenmitte sehen, beträgt die Größe des linearisierten Plasmids 4361 Basenpaare. Bevor Sie mit einem Plasmid arbeiten, ist es ratsam, es durch Schneiden mit einem eindeutigen Restriktionsenzym zu linearisieren, um zu überprüfen, ob die angegebene Größe ungefähr der erwarteten entspricht.
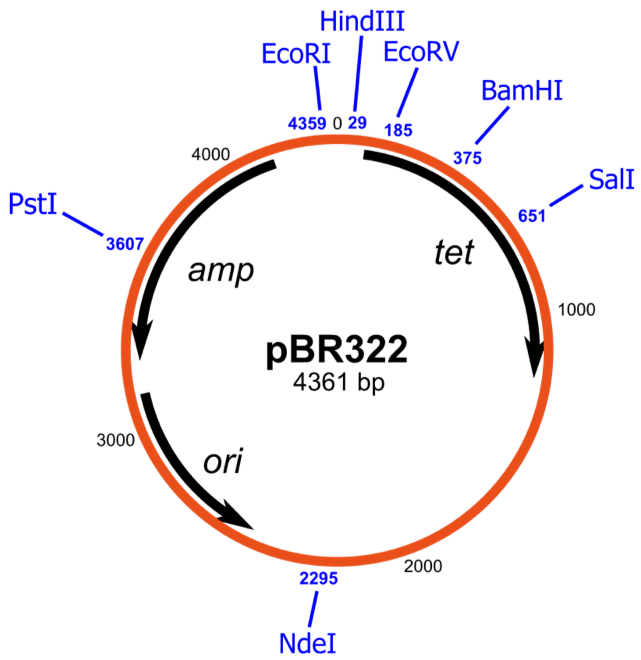
Abbildung 1. Schaltplan von pBR322. Bild von Avacop & Yikrazuul via Wikimedia Commons
Die schwarzen Pfeile zeigen die Richtung der Transkription, die für das Klonen wesentlich ist. Wenn Sie Ihr interessierendes Gen in der Mitte eines anderen Gens klonen, stellen Sie sicher, dass beide in die gleiche Richtung transkribiert werden. Andernfalls kann der native Promotor Ihre Genexpression stören.
Was bedeutet „Ori“?
In Gensequenzdatenbanken, wie Entrez-PubMed, werden Plasmidsequenzen ausgehend vom ori als lineare Sequenzen dargestellt. „Ori“ bedeutet den Ursprung der Plasmidreplikation. Was auch immer du tust, ändere es nicht! Sobald ein Plasmid nicht in der Lage ist, sich zu replizieren, ist es nutzlos.
Die andere Sache, an die man sich bei ori erinnern sollte, ist, dass Plasmide mit demselben Ursprung oft inkompatibel sind. Dies bedeutet, dass Sie nicht in der Lage sein werden, zwei von pBR323 abgeleitete Vektoren in einer Zelle zu erhalten, selbst wenn sie Gene für unterschiedliche Antibiotikaresistenzen aufweisen. Das pBR322-Ori wird auch in pUC18 verwendet, dem zweithäufigsten in eukaryotischen Vektoren verwendeten Rückgrat.
Wo finde ich die Restriktionsstellen?
Restriktionsstellen für entsprechende Enzyme sind als vertikale Linien mit der Position der Ausgangsnukleotide dargestellt. Die Sites sollten eindeutig sein, aber es lohnt sich zu überprüfen, da abgeleitete Vektoren oft zusätzliche vergessene Sequenzen enthalten.
Was ist mit Antibiotikaresistenzgenen?
pBR322 hat zwei Antibiotikaresistenzgene: tet (Tetracyclinresistenz) und amp (Ampicillinresistenz). Diese Gene kodieren eine Effluxpumpe (tetR) und Beta-Lactamase (ampR), um Tetracyclin bzw. Ampicillin aus der Zelle auszuscheiden. Tet und amp werden in verschiedene Richtungen gelesen.
Bedenken Sie, dass das Enzym Beta-Lactamase bei der Entgiftung von Penicillin-Antibiotika nicht spezifisch ist. Selbst wenn Sie also zwei Plasmide mit unterschiedlichen Replikationsursprüngen haben, können Sie nicht zwei Plasmide gleichzeitig auswählen, wenn eines ein Methicillin-Resistenzgen und das andere ein Ampicillin-Resistenzgen exprimiert.
Wenn Sie Restriktionsenzymstellen verwenden, um Ihr interessierendes Gen in Ihr Plasmid zu klonen, achten Sie darauf, welche Stellen in Ihr Antibiotikaresistenzgen fallen. Zum Beispiel schneidet PvuI in der Mitte von AmpR und BamHI in der Mitte von TetR. Und wie wir alle wissen, führt die Störung in einem Gen zur Inaktivierung der Genfunktion – in diesem Fall zur Antibiotikaresistenz.
Wie startet und stoppt die Replikation?
Plasmide enthalten neben Genen häufig Transkriptionspromotoren und Terminatoren, die von E. coli-Phagen abgeleitet sind. Promotoren aus den Phagen SP6 und T7 werden häufig für die In-vitro-RNA-Amplifikation verwendet. Sie benötigen Phagenpolymerasen und sind daher in vivo inaktiv.
Unten ist eine Karte von pTLNX , einem Xenopus-Oozyten-Expressionsvektor (Abbildung 2). Neben dem bekannten pBR322-Ursprung und den Antibiotikaresistenzgenen AmpR und CmR (Chloramphenicol-Resistenz) sind auch SP6- und lacUV-Promotoren vorhanden. Nach dem SP6-Promotor ermöglicht der rrnBT2-Terminator eine effiziente Terminierung von Genen, die in die multiple Klonierungsstelle klonieren2 (Abbildung 2).
Der pTLNX-Vektor hat auch ein Gen für Plasmidselektion (ccdB), zusammen mit Virus SV40 Kernlokalisierungssignal und Xenopus globine 3′ UTR, das hohe Expressionsniveaus von klonierten Genen ermöglicht.
Kennen Sie Ihre Quellen
Hausgemachte Karten sind oft unzuverlässig, da sie „unwichtige“ Funktionen auslassen, die für Ihr Experiment kritisch sein können. Wenn Sie eine Vektorkarte benötigen, ist es besser, bekannte Karten-Repositorys wie Addgene, Entrez-PubMed und Websites von Unternehmen zu verwenden, die Ihren Vektor von Interesse verkaufen.

Abbildung 2. Schaltplan von pTLNX. Bild von Addgene.
Plasmidkarten entwickeln sich ständig weiter, so dass es wahrscheinlich ist, dass Ihre Beiträge für Ihre zukünftigen Kollegen übrig bleiben. Bitte geben Sie so viele Details wie möglich in Ihre Karte ein! Glückliche Karte lesen und Zeichnen.
Quellen:
- Balbás P, Soberón X, Merino E, Zurita M, Lomeli H, Valle F, Flores N, Bolivar F (1986). „Plasmidvektor pBR322 und seine speziellen Derivate – eine Überprüfung“. Gen. 50 (1-3): 3–40. doi:1016/0378-1119(86)90307-0.
- Geertsma ER, Dutzler R. (2011). Ein vielseitiges und effizientes Hochdurchsatz-Klonierungswerkzeug für die Strukturbiologie. 50(15):3272-8. ust-idnr.: 10.1021/bi200178z
Hat dir das geholfen? Dann teilen Sie bitte mit Ihrem Netzwerk.